Schritt für Schritt verfolgt
ATP-Spaltung in Membranprotein erstmals dynamisch gemessen
RUB-Forscher berichten im Journal of Biological Chemistry
Bochum, 09.07.2012
Presseinformation 242/2012
Wie ein Transportprotein aus dem Energiespeichermolekül ATP seine Antriebskraft gewinnt, haben RUB-Forscher dynamisch verfolgt. Mit der zeitaufgelösten Infrarotspektroskopie beobachteten sie die strukturellen Veränderungen in dem bakteriellen Membranprotein MsbA und seinem Interaktionspartner ATP. Über die Ergebnisse berichten die Forscher um Prof. Dr. Eckhard Hofmann und Prof. Dr. Klaus Gerwert vom Lehrstuhl Biophysik in der aktuellen Ausgabe des Journal of Biological Chemistry.
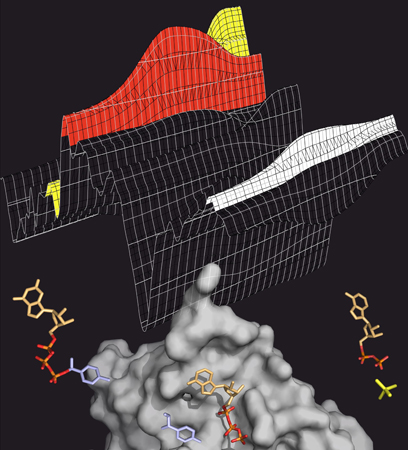
ATP-Spaltung: Das Transportprotein MsbA (grau) spaltet ATP (bunt), um Energie für den Transportprozess zu gewinnen. ATP besitzt drei Phosphatgruppen (orange-rot). Wird eine davon abgespalten (gelb), wird Energie frei. Den Spaltprozess kann man im Infrarot-Spektrum verfolgen (oben), in dem die verschiedenen ATP-Zwischenprodukte charakteristische Banden hinterlassen (rot: ATP, gelb: abgespaltenes Phosphat, weiß: Protein).
© Falk Syberg
|
Transportproteine sind mit verschiedenen Krankheiten assoziiert
ABC-Transporter sind Membranproteine, die verschiedene Substanzen von einer Seite der Zellmembran auf die andere befördern. Die Antriebskraft dafür liefert das Molekül ATP, ein universeller Energiespeicher der Zellen. ATP besitzt drei Phosphatgruppen; wird eine davon abgespalten, wird Energie frei. Die Transporter sind medizinisch relevant, da sie bei der Multiresistenz von Krebszellen gegen Chemotherapeutika eine zentrale Rolle spielen und mit verschiedenen Erbkrankheiten wie Mukoviszidose zusammenhängen. In den letzten Jahren haben Forscher die 3D-Strukturen von mehreren dieser Transporter auf atomarer Ebene aufgeklärt. Obwohl die Architektur der Nanomaschinen bekannt ist, fehlt bisher das detaillierte Verständnis darüber, wie die Spaltung des Energieträgers ATP dynamisch den Transport von verschiedenen Substanzen über biologische Membranen ermöglicht.
Protein steuert ATP-Spaltung
In dem Fetttransporter MsbA aus dem Bakterium Escherichia coli verfolgten die Bochumer Forscher nun erstmals dynamisch die ATP-Spaltung, Hydrolyse genannt. Mit der Fourier-Transform-Infrarotspektroskopie untersuchten sie die Motordomäne von MsbA, also den Teil des Proteins, an dem sich die ATP-Spaltung vollzieht. Mit der Methode können Forscher kleinste Veränderungen im Protein im Bereich von Nanosekunden verfolgen. Gleichzeitig erfasst das Verfahren aber auch Veränderungen in den Molekülen, mit denen das Protein interagiert – in diesem Fall ATP.
Phosphatsignale verraten, was bei der Spaltung vor sich geht
Die große Herausforderung bei der Datenanalyse ist es, die Signale im gemessenen Spektrum bestimmten Molekülen bzw. Molekülgruppen zuzuordnen. Gelingt das, kann man erkennen, welche Molekülgruppen sich zu welchem Zeitpunkt strukturell verändern. Die Biophysiker markierten die Phosphatgruppen des ATP-Moleküls, so dass sie charakteristische Signale im Spektrum hinterließen. Auf diese Weise verfolgten sie, wie ATP an das Transportprotein band, eine seiner drei Phosphatgruppen abgespalten und in die Umgebung abgegeben wurde, ohne sich zuvor noch einmal an das Protein anzuheften. "Unsere Daten liefern auch wichtige Hinweise dafür, wie sich das Protein während der ATP-Hydrolyse bewegt. Das legt den Grundstein für die Untersuchung des gesamten Membranproteins, die wir als nächstes in Angriff nehmen", so Prof. Hofmann. Gefördert wurden die Untersuchungen vom Protein Research Department der RUB und aus Mitteln des SFB 642 "GTP- und ATP-abhängige Membranprozesse", deren Sprecher Prof. Gerwert ist.
Titelaufnahme
F. Syberg, Y. Suveyzdis, C. Kötting, K. Gerwert, E. Hofmann (2012): Time-resolved fourier transform infrared spectroscopy of the nucleotide-binding domain from the ATP-binding cassette transporter MsbA. ATP Hydrolysis is the rate-limiting step in the catalytic cycle, Journal of Biological Chemistry, doi: 10.1074/jbc.M112.359208
Weitere Informationen:
Prof. Dr. Eckhard Hofmann
AG Proteinkristallographie
Fakultät für Biologie und Biotechnologie der Ruhr-Universität
44780 Bochum
Tel: 0234/32-24463
E-Mail: eckhard.hofmann@bph.rub.de
